Forschungsprojekte Abt. Technische Enzymologie
Forschungsschwerpunkte
Die Abteilung Technische Enzymologie beschäftigt sich mit Aufgaben, die für die Anwendung von Enzymen als Biokatalysatoren von grundlegender Bedeutung sind.
Im Mittelpunkt der Untersuchungen stehen gegenwärtig die Themenkomplexe
1. Mechanismen der Proteinstabilisierung
2. Phospholipasen und Phospholipidtransformationen
3. Ribonucleasen als potentielle Tumortherapeutika
1. Mechanismen der Proteinstabilisierung
Um Enzyme zur Katalyse chemischer Synthesen nutzen zu können, bedarf es oft einer Stabilisierung ihrer natürlichen Struktur. Auf mehr oder weniger empirischem Wege sind hierfür eine Vielzahl von Möglichkeiten gefunden worden, die entweder auf chemischen bzw. gentechnischen Veränderungen der Proteinstruktur oder auf Veränderungen der Mediumzusammensetzung beruhen, ohne dass sich daraus verallgemeinerungsfähige Strategien ableiten lassen.
Studien zu den Stabilisierungsmechanismen von Proteinen in unserem Labor haben zu der Hypothese geführt, dass für die gezielte Stabilisierung eines Proteinmoleküls bestimmte Regionen der Oberfläche, insbesondere Strukturloops, eine entscheidende Rolle spielen. Gelingt es, diese sog. Entfaltungsregionen zu lokalisieren, können sie durch gerichtete Immobilisierung, die Einführung von Disulfidbrücken (Abb. 1) oder andere chemische bzw. gentechnische Methoden fixiert werden, was eine entscheidende Stabilisierung des Proteinmoleküls zur Folge hat. An verschiedenen Proteinen (Ribonucleasen, neutralen Proteasen, Barstar, Phospholipasen) bzw. ihren lokal modifizierten Varianten verfolgen wir die Verifizierung des Modells der Enfaltungsregionen, indem Entfaltungsereignisse auf submolekularer Ebene mit Hilfe von limitierter Proteolyse, Stopped-Flow-Fluoreszenz- und CD-Spektroskopie, NMR-Spektroskopie oder Einzelmolekülspektroskopie analysiert werden.
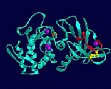
Abb. 1: Modell der neutralen Protease aus Bacillus stearothermophilus.
Die Entfaltungsregion ist rot gekennzeichnet, die Disulfidbrücke ist gelb markiert, die Calcium-Ionen sind violett, das Zink-Ion ist orange dargestellt.
2. Phospholipasen und Phospholipidtransformationen
Die in der Natur ubiquitär vorkommenden Phospholipasen (PLA1, PLA2, PLC und PLD) katalysieren spezifisch die Spaltung der vier Esterbindungen in Glycerophospholipiden (Abb. 2). In der Natur haben sie vielfältige Funktionen inne, vom Membranabbau und –umbau bis zur Zellregulation und Signalübertragung. In der Industrie und in chemischen Labors werden Phospholipasen, insbesondere PLA2 und PLD, zur Biotransformation von Phospholipiden genutzt.
In unserer Abteilung wurden die molekularbiologischen Grundlagen zur rekombinanten Gewinnung verschiedener Phospholipasen, insbesondere pflanzlichen Ursprungs, erarbeitet, um die Enzyme gezielt für die Anwendung modifizieren zu können. Zu diesen Untersuchungsobjekten gehören PLD-Isoenzyme aus Weißkohl bzw. Schlafmohn sowie PLA2 aus der Honigbiene und aus Arabidopsis thaliana (Abb. 3). Darüber hinaus befassen wir uns mit den Enzym-Phospholipid-Wechselwirkungen bei der Aktivierung und Katalyse der Phospholipasen in wässrigen und nichtwässrigen Reaktionssystemen und nutzen die Erkenntnisse zur Synthese von neuen Phospholipiden bzw. Phospholipidanaloga.
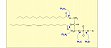
Abb. 2: Angriffsorte der Phospholipasen an einem Glycerophospholipidmolekül. PLA1: Phospholipase A1; PLA2: Phospholipase A2; PLC: Phospholipase C; PLD: Phospholipase D
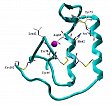
Abb. 3: Strukturmodell der Phospholipase A2 aus Arabidopsis thaliana. Das Modell umfasst die Aminosäurereste Tyr35 - Cys102. Die Kugel in Violett symbolisiert das für die Aktivität essentielle Calcium-Ion.
3. Ribonucleasen als potentielle Tumortherapeutika
Die Zytotoxizität von Ribonucleasen zur Krebstherapie zu nutzen, ist ein in jüngster Zeit verfolgter neuer Ansatz in der Tumorforschung. Er basiert vor allem auf den erfolgversprechenden bereits im klinischen Stadium befindlichen Versuchen mit Onconase, einer Ribonuclease aus dem Nördlichen Leopardfrosch. Ihre hohe Wirksamkeit wird vor allem ihrer geringen Bindungsaffinität zu dem humanen zellulären Ribonuclease-Inhibitor zugeschrieben. In der Arbeitsgruppe wird an einem neuen Konzept zur Entwicklung von Ribonuclease-Konstrukten mit zytotoxischer Wirkung gearbeitet, das auf der gentechnischen Verknüpfung von zwei Säuger-Ribonuclease-Molekülen über einen Peptidlinker beruht. Aufgrund sterischer Hinderung sollten diese Konstrukte jeweils nur ein Molekül des Inhibitors binden können (Abb. 4), während das zweite Molekül des Ribonuclease-Tandems noch zytotoxische Aktivität entwickeln kann. Unsere Studien zeigten dabei, dass die Effizienz der Aufnahme der Ribonucleasen in die Zielzelle offenbar von größerer Bedeutung ist als die Inhibitorbindung.

Abb. 4: Modell des Ribonuclease A-Tandemenzyms im Komplex mit einem Inhibitormolekül
Das Inhibitormolekül (in Grün) umschließt eine der Ribonuclease A-Einheiten, während die zweite Einheit frei bleibt. Die α-Helix- bzw. β-Faltblattstrukturen der beiden Ribonuclease-Moleküle sind rot bzw. gelb markiert.


